-
抗体試薬
- フローサイトメトリー用試薬
-
ウェスタンブロッティング抗体試薬
- イムノアッセイ試薬
-
シングルセル試薬
- BD® OMICS-Guard Sample Preservation Buffer
- BD® OMICS-One Protein Panels
- BD® AbSeq Assay
- BD® Single-Cell Multiplexing Kit
- BD Rhapsody™ ATAC-Seq Assays
- BD Rhapsody™ Whole Transcriptome Analysis (WTA) Amplification Kit
- BD Rhapsody™ TCR/BCR Next Multiomic Assays
- BD Rhapsody™ Targeted mRNA Kits
- BD Rhapsody™ Accessory Kits
- BD® OMICS-One Immune Profiler Protein Panel
- BD OMICS-One™ WTA Next Assay
-
細胞機能評価のための試薬
-
顕微鏡・イメージング用試薬
-
細胞調製・分離試薬
Old Browser
Looks like you're visiting us from {countryName}.
Would you like to stay on the current location site or be switched to your location?
Overview
シングルセルレベルでオープンクロマチン領域をマッピングすることにより、個々の細胞における運命決定や刺激への反応などエピゲノム制御のランドスケープが明らかになります。
Tn5 TransposaseとNGSを利用してオープンクロマチン領域を決定する手法 (ATAC-seq) は、従来の手法よりも高感度で偏りのないゲノム全体のクロマチンアクセシビリティを読み取ることを可能にします。
同じ細胞からのシングルセルトランスクリプトームデータと組み合わせて得られるマルチオミックなデータにより、遺伝子制御クロマチンの状態と各細胞の遺伝子発現データとの直接的な関連付けが可能になり、さまざまな条件での細胞状態に関するメカニズムの理解をさらに深めることができます。
BD Rhapsody™ ATAC-Seq Assayにより、シングルセルから再現性の高いオープンクロマチンプロファイリングデータを得られるだけでなく、オープンクロマチン領域とトランスクリプトームのマルチオミクス解析データを1度の実験で得ることが可能です。
BD Rhapsody™ ATAC-Seq Assay のカタログ で詳細をご覧ください。
Features
新開発のBD Rhapsody™ Enhanced Cell Capture Beads V3で検証されたBD Rhapsody™ ATAC-Seq Assayは、さまざまなサンプルやユーザー、幅広い細胞インプットにわたって一貫した高品質のデータを生成します。主な特徴は次のとおりです。
- さまざまな実験条件にわたって高い感度と特異性
- 幅広い細胞インプットに対応可能
- 同一細胞におけるエピゲノムとトランスクリプトームの統合的な特性評価
- カスタムBD® Nuclear Antibody-Oligonucleotide Conjugates との互換性
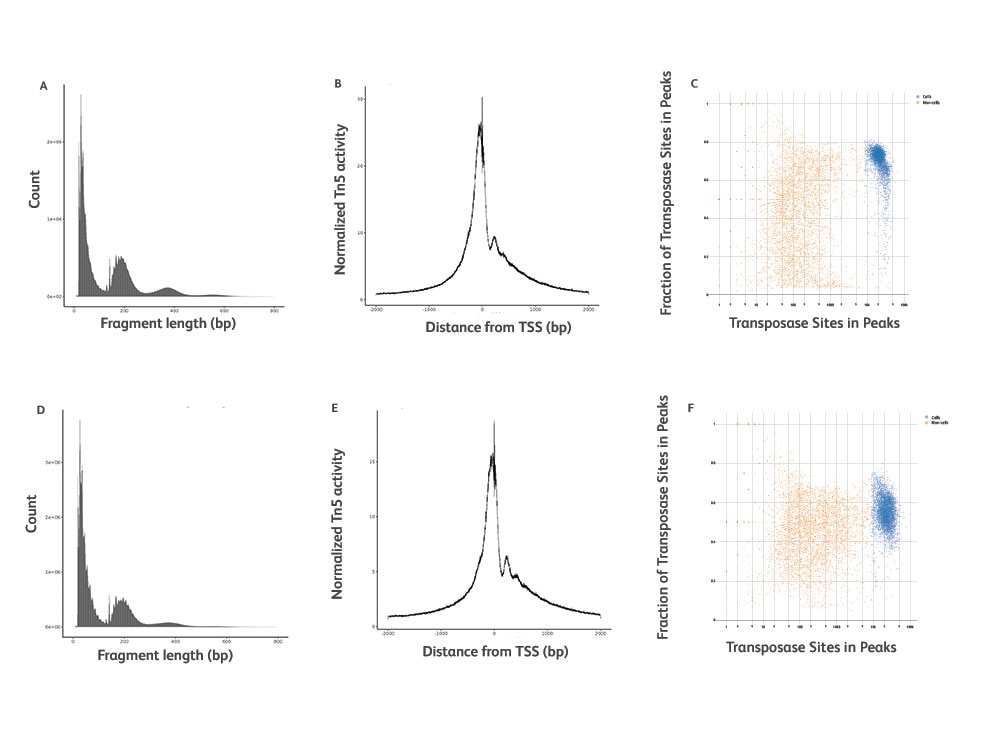
エピゲノムの不均一性を高精度で明らかに

BD Rhapsody™ シングルセル ATAC-Seq Assayの感度と特異性に関するメトリクス
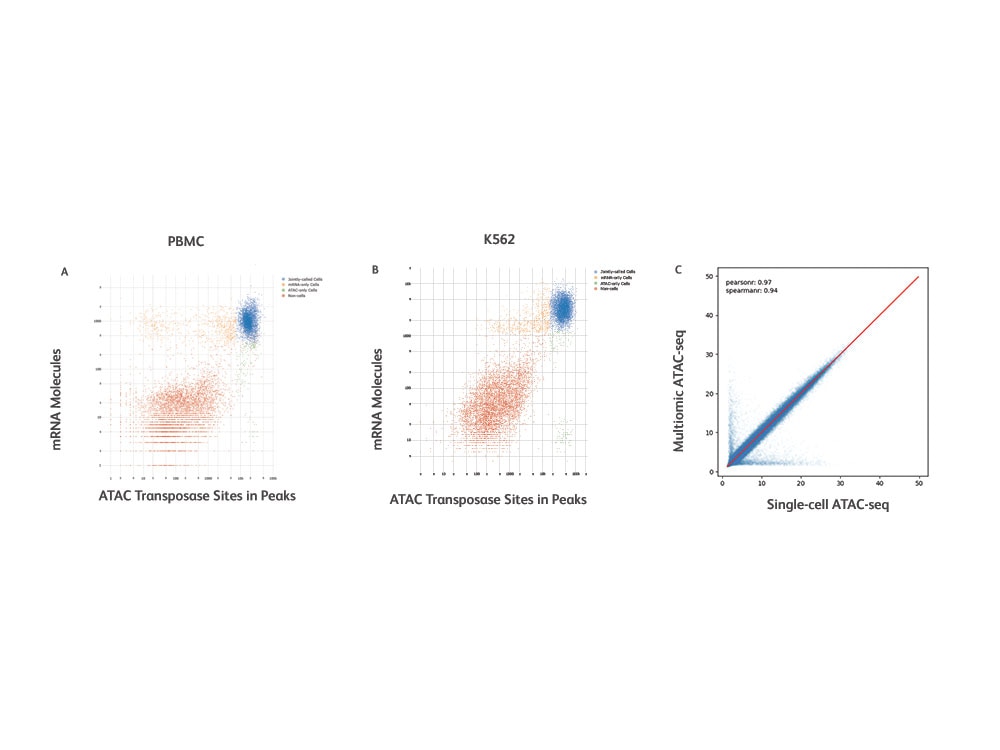
シングルセルレベルでの遺伝子制御と発現の関係を解明

BD Rhapsody™ シングルセル マルチオミック ATAC-Seq Assayの感度と特異性に関するメトリクス
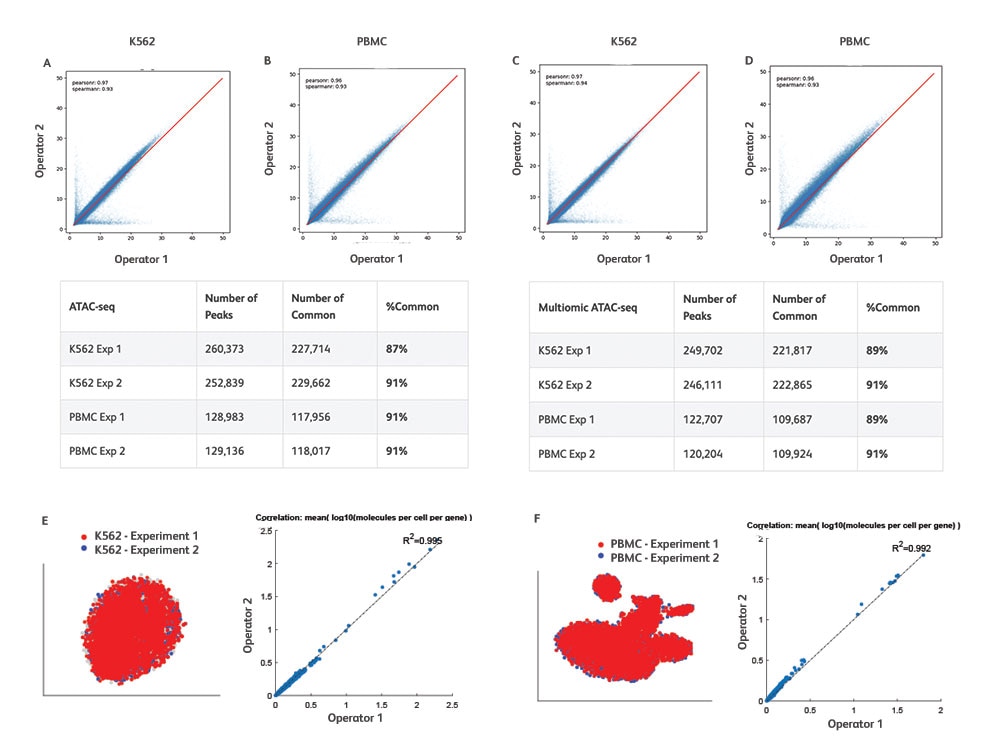
異なるサンプルや実験間で再現可能な結果

BD Rhapsody™ ATAC-Seq Assayによる高い再現性
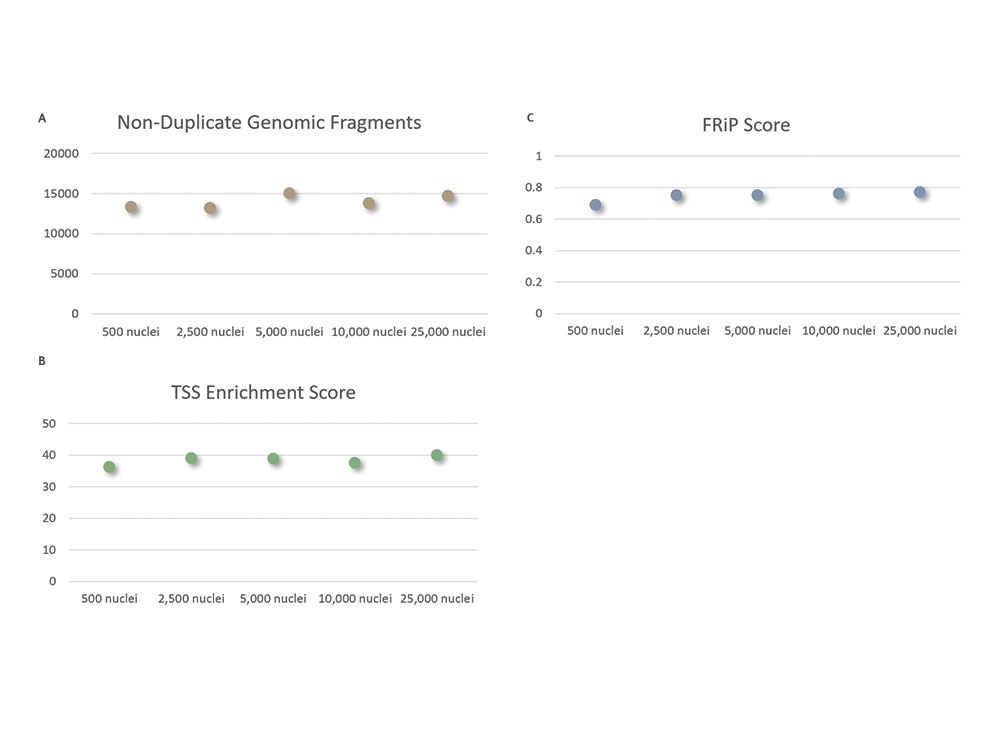
幅広い細胞数のインプットにわたって一貫性のある高品質なデータを生成

さまざまな細胞のインプットにわたって安定したパフォーマンスを実現します。
Applications
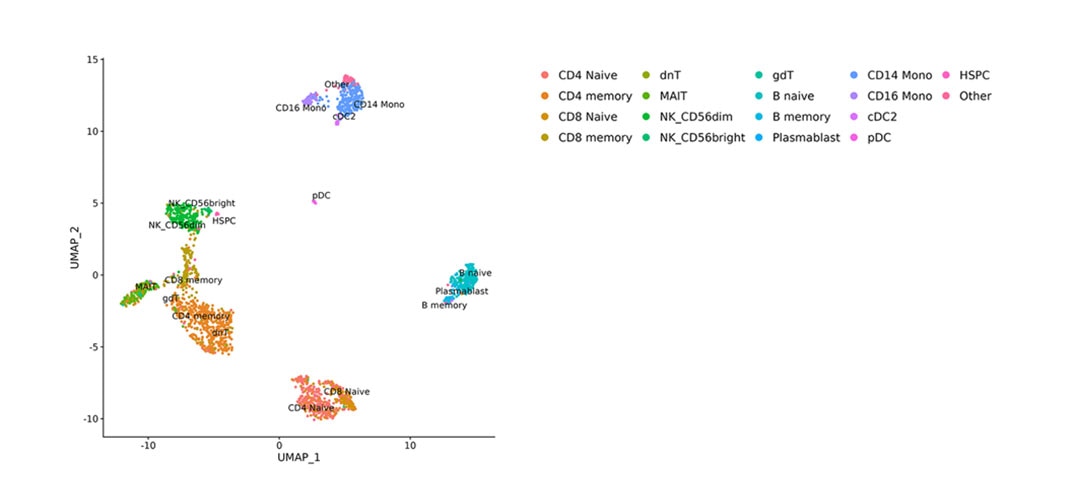
In this study, the BD Rhapsody™ Single-Cell Multiomic ATAC-Seq Assay was used to analyze 2,190 nuclei from human peripheral blood mononuclear cells (PBMCs). A joint whole transcriptome analysis (WTA) and ATAC-seq dimensionality reduction was performed by Uniform Manifold Approximation and Projection (UMAP) and cells were annotated as members of distinct cell types using the WTA data from a PBMC reference atlas.
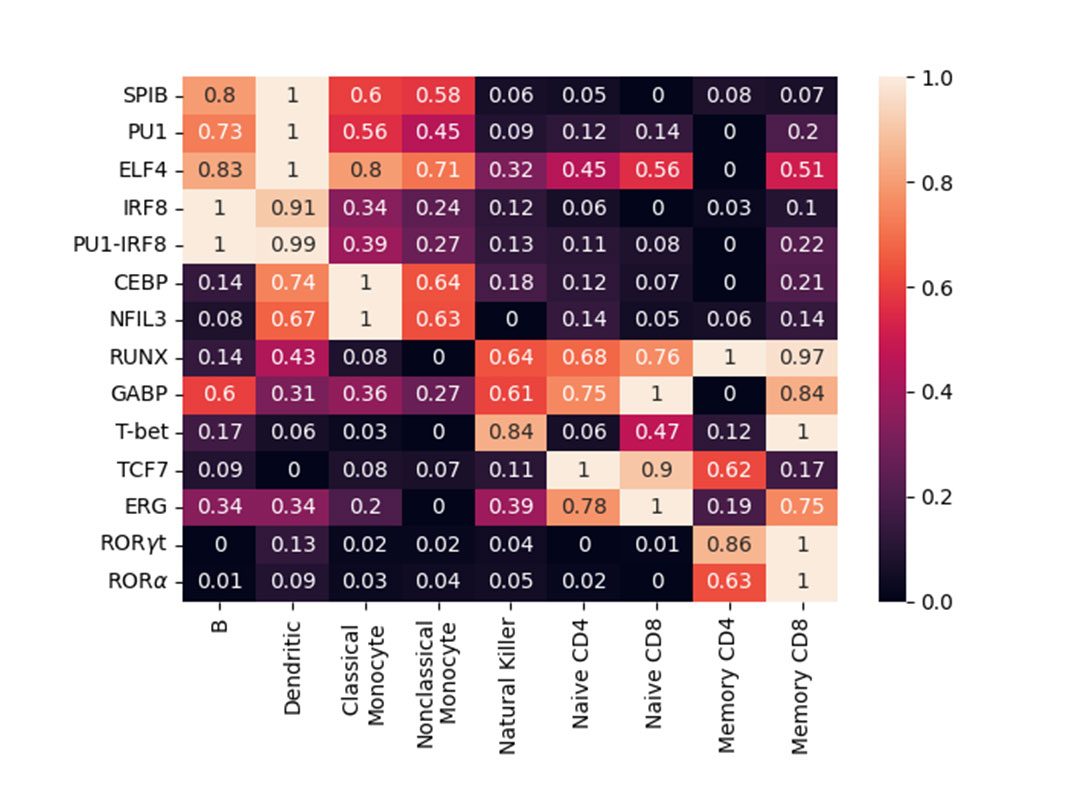
The heat map shows normalized enrichment scores of cell type–specific transcription factor motifs. Motif scores were calculated using a binomial distribution, determining the relative enrichment of each motif in differentially accessible regions of a given cell type compared to GC-matched background regions. The scores were then normalized across cell types per motif on a 0–1 scale, where 0 indicates least enrichment and 1 indicates highest enrichment of each motif.
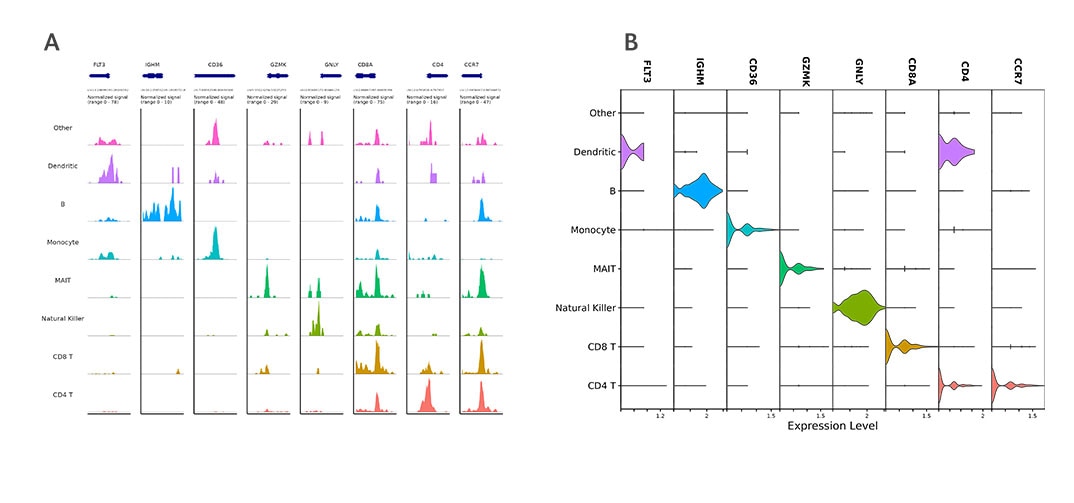
A. Read density across each ATAC-seq cluster at the transcription start sites of cell type marker genes.
B. Violin plots showing cell type-specific gene expression in WTA data.
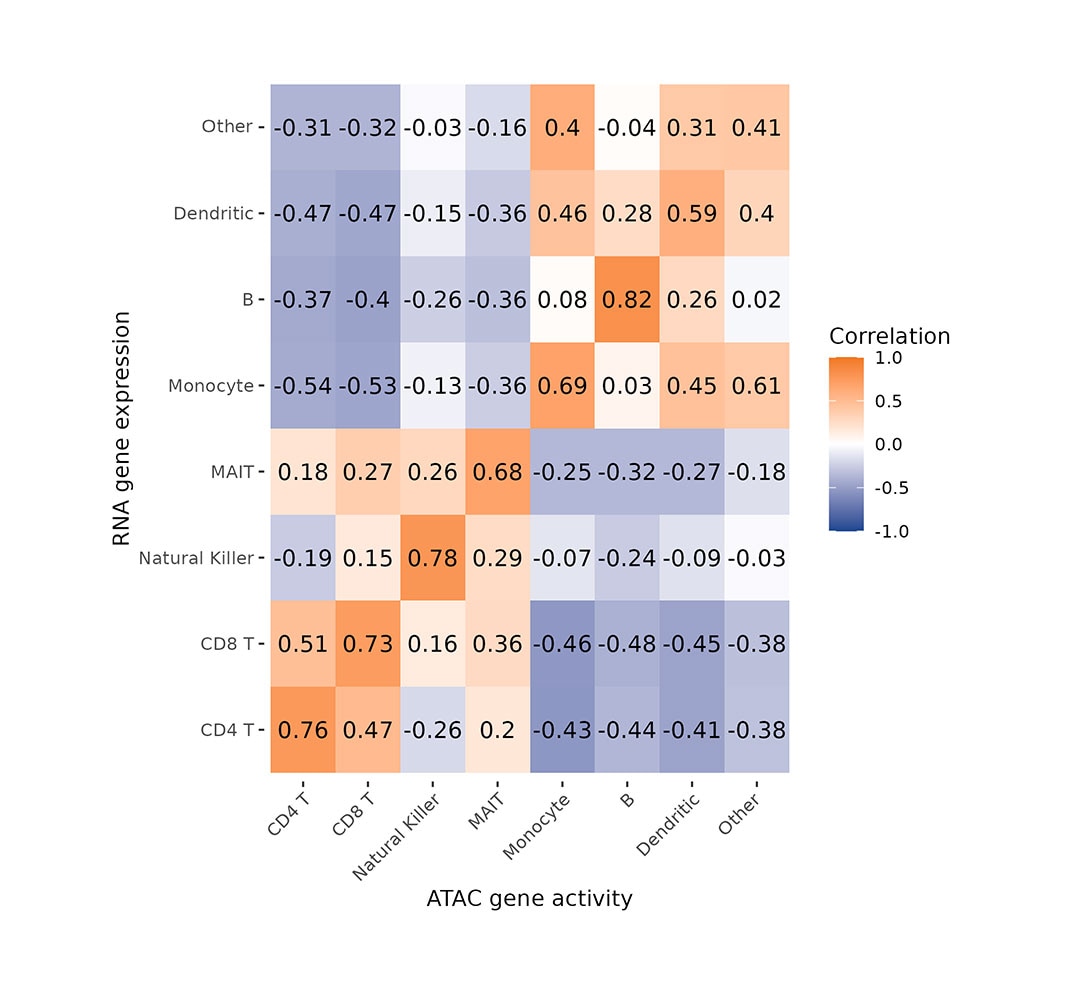
This heatmap shows Pearson’s correlation coefficients between ATAC-seq gene activity scores and gene expression values in PBMCs, with each row representing a cell type in WTA data and each column a cell type in ATAC-seq data.
研究用です。本製品は、疾病の診断・治療または予防に使用することはできません。
