-
抗体試薬
- フローサイトメトリー用試薬
-
ウェスタンブロッティング抗体試薬
- イムノアッセイ試薬
-
シングルセル試薬
- BD® OMICS-Guard Sample Preservation Buffer
- BD® OMICS-One Protein Panels
- BD® AbSeq Assay
- BD® Single-Cell Multiplexing Kit
- BD Rhapsody™ ATAC-Seq Assays
- BD Rhapsody™ Whole Transcriptome Analysis (WTA) Amplification Kit
- BD Rhapsody™ TCR/BCR Next Multiomic Assays
- BD Rhapsody™ Targeted mRNA Kits
- BD Rhapsody™ Accessory Kits
- BD® OMICS-One Immune Profiler Protein Panel
- BD OMICS-One™ WTA Next Assay
-
細胞機能評価のための試薬
-
顕微鏡・イメージング用試薬
-
細胞調製・分離試薬
Old Browser
Looks like you're visiting us from {countryName}.
Would you like to stay on the current location site or be switched to your location?
Single-Cell Multiomics
シングルセル マルチオミクス (scM) は、細胞生物学を理解し、精査する方法に革命をもたらすと期待される最先端のアプローチです。マルチオミクス、つまりゲノム、トランスクリプトミクス、プロテオーム、その他のオミクス データを統合する機能は、最初は冗長に思えるかもしれません。理論的には1つのオミクスですべての分子情報を取得できるのに、なぜ複数のレイヤーの分子情報を統合する手間をかけるのでしょうか?その答えは生物学的ネットワークの複雑さにあります。それぞれのオミクスの解析結果は重要な洞察を提供しますが、単一の「オミクス」では細胞プロセスの複雑なネットワークを完全に解明することはできません。たとえば、DNAは細胞の潜在能力を決定しますが、すべての遺伝子がRNAに転写されるわけではなく、すべてのRNAがタンパク質に翻訳されるわけではありません。各レイヤーの統合的な解析は、細胞の全体的な機能に関する詳細な情報を提供します。
マルチオミクスが今なぜ重要なのか?
マルチオミクス解析は生物学的サンプルに対してより頑健な分類手法を提供し、例えばがんのサブタイプの特定や患者の層別化などのタスクに役立ちます。マルチオミクス解析により、遺伝子型と表現型の関係をより深く理解し、生物学的特性や疾患の根底にある基本的なメカニズムに関する洞察を得られます。
2019年にシングルセルマルチモーダルオミクスが Nature Method of the Year in 2019, に選ばれたことは、その重要性を認識し、生物学研究の進展におけるその中心的な役割を強調しています。
シングルセル マルチオミクスの領域をさらに深く探求するにつれて、生物学の理解がさらに進み、個別化医療、疾患治療などにおける画期的な進歩への道が開かれます。
シングルセルマルチオミクス機器、scM試薬、scMインフォマティクス分析ソリューションについては、BDツールをご覧ください。
The Five Omes
Genomics
ゲノミクスは生物のゲノムを構成するヌクレオチド塩基の配列を解読することによって生物のDNAを研究する学問です。研究者は遺伝的変異、遺伝、進化的関係についての貴重な洞察を得ることができます。

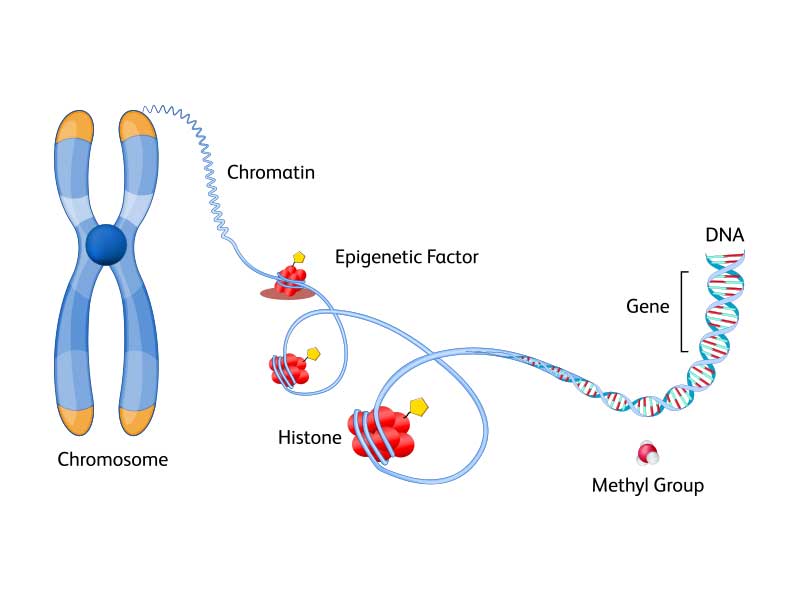
Epigenomics
エピゲノミクスは、遺伝子コードを変えることなく、遺伝子発現を調節するDNAおよび関連タンパク質への動的な修飾を探求します。DNAメチル化、ヒストン修飾、非コードRNA調節などのエピジェネティックなメカニズムは、細胞の分化、発生、および環境刺激への応答を調整する上で重要な役割を果たします。
シングルセルエピゲノミクスにおけるBDのソリューションをご覧ください。
Transcriptomics
トランスクリプトミクスは、細胞または組織内のRNA分子の広大な領域を詳しく調べ、さまざまな条件下での遺伝子発現パターンの動的なレパートリーを捉えます。
WTA(全トランスクリプトーム)またはTargeted mRNAのシングルセル トランスクリプトミクスのためのBDのソリューションをご覧ください。
Proteomics
タンパク質は細胞の働き手であり、触媒作用やシグナル伝達から構造的支持や防御に至るまで、多様な機能を実行します。プロテオミクス解析は、タンパク質の量、翻訳後修飾、およびタンパク質間相互作用に関する洞察を提供します。
細胞表面タンパク質の単一細胞プロテオミクス そして 細胞表面タンパク質および細胞内タンパク質のシングルセル プロテオミクスに関するBDのソリューションをご覧ください
い。 BDのソリューションをご覧ください。
Metabolomics
メタボロミクスは、細胞代謝の構成要素として機能する低分子代謝物の研究です。代謝物には、アミノ酸、脂質、糖、神経伝達物質など、多様な化合物が含まれます。メタボロミクスは、生理的状態、環境曝露、および疾患表現型に関連する代謝経路、代謝フラックス、および代謝シグネチャーを解明します。
弊社のブログページで、シングルセル マルチオミクスの最新動向について詳しくご覧ください。

生物学の分野を進歩させるために、研究者が画期的な研究でシングルセル マルチオミクスをどのように活用しているかをご覧ください。
Immuno-Oncology
1. 慢性リンパ性白血病 (CLL) - この研究は、CLL の複雑性と不均一性を解明する上でのシングルセル マルチオミクスの有効性を実証し、その病因を理解し、治療戦略に情報を提供するための貴重な洞察を提供します。データシート「Exploring tumor heterogeneity of chronic lymphocytic leukemia using single cell multiomics.」をご覧ください。
2. B細胞リンパ腫 - この研究では、B細胞リンパ腫の腫瘍微小環境における悪性B細胞と免疫細胞の相互作用を詳しく調べ、この疾患の治療標的を特定するための洞察を提供します。データシート「Leveraging the power of high-parameter cell sorting and single-cell multiomics to profile intratumoral immune cells in a model of B-cell lymphoma.」をご覧ください。
3. メラノーマ - この研究は、がん関連造血における腫瘍誘発性の変動を調査し、腫瘍が異なる造血幹細胞および前駆細胞(HSPC)集団にどのように影響するかに焦点を当てています。データシート「Assessing tumor-driven perturbations during hematopoiesis in a melanoma mouse model through single-cell multiomic analysis.」をご覧ください。
4. 大腸がん — 『Cell Reports Medicine』誌に掲載された『Human tumor-infiltrating MAIT cells display hallmarks of bacterial antigen recognition in colorectal cancer』という研究では、大腸がんにおいて微生物抗原に応答する特定の腫瘍浸潤MAIT細胞のサブセットを発見しました。これらの細胞を理解することは、がん治療のためにそれらやマイクロバイオームを操作する道を開く可能性があります。」
Download our application ebook, Advancing immuno-oncology through single-cell multiomics, to see how single-cell multiomics, the BD Rhapsody™ System and cell sorting can be used to gain important insights into cancer immunology.
Autoimmunity
- 全身性エリテマトーデス - “Chronic immune activation in systemic lupus erythematosus and the autoimmune PTPN22 Trp620 risk allele drive the expansion of FOXP3+ regulatory T cells and PD-1 expression”では、SLE患者はPD-1、IL-2、可溶性SIGLEC-11に関連する胸腺由来Tregの増殖により、CD4+FOXP3+細胞が多くなっていることが判明しました。2
- 関節リウマチ (RA) - Nature Medicine の論文 “Distinct synovial tissue macrophage subsets regulate inflammation and remission in rheumatoid arthritis”「では、RAの持続的な寛解の細胞および分子メカニズムについて詳しく説明しています。この研究では、炎症と寛解を制御する異なる滑膜組織マクロファージサブセットに焦点を当て、RAにおける炎症の回復を促進し、滑膜の恒常性を回復するための潜在的な治療標的を明らかにしました。3
- 自己免疫性脱髄 - PNASに掲載された研究では、TNF-αシグナル伝達が末梢神経の炎症と自己免疫に非常に重要であることが明らかになりました。Aire 欠損マウスでは TNF-αシグナル伝達が増加し、免疫細胞が動員されて活性化し、自己免疫性脱髄が悪化します。TNF-αまたはその受容体を阻害すると神経障害が改善するため、抗TNF-α療法がGBSやCIDPなどの炎症性神経障害を治療できる可能性があることが示唆されています。4
- 1型糖尿病(T1DM) - この研究では、シングルセル VDJシーケンシングを使用して日本人のT細胞受容体 (TCR) の多様性を調査し、自己反応性T細胞において異なる TCR配列と、健康な被験者と比較したCD8+ および FOXP3+ 細胞の遺伝子発現の違いを示し、T1DMにおけるTCRの多様性と免疫応答の変化を示唆しました。5

Infectious Diseases
- 急性中耳炎 - 急性中耳炎のラットモデルにおいて、シングルセル トランスクリプトーム プロファイリングにより、貪食作用に関連する二重機能を持つ細胞が明らかになりました。これらの細胞は専門的な貪食細胞と共存し、炎症の進行に影響を与えました。マクロファージは炎症反応において中心的な役割を果たし、M2型への分極は免疫微小環境に影響を及ぼします。6
- COVID-19 - この研究では、合併症のないCOVID-19回復者の単球における長期的な免疫学的瘢痕としてHIF1Aが特定されました。これは疾患の重症度と相関しており、特に症状のない回復期において、COVID-19後の免疫モニタリングと治療戦略設計のためのバイオマーカーとしての可能性を示唆しています。7
- 敗血症 - この研究では、敗血症時のマクロファージにおけるミトコンドリアSTAT3の役割を調べました。ミトコンドリアSTAT3は、USP501を媒介されるCPT1aの安定化を通して脂肪酸酸化(FAO)を促進し、敗血症を悪化させることが明らかになりました。8
- 結核 - この研究では、潜在性結核ドナーからのγδ T細胞を濃縮し、シングルセル免疫プロファイリングを使用して解析しました。この研究では、結核に対する免疫反応を理解する上で極めて重要な、完全長 γδ T 細胞受容体配列の特定とクローン増殖に焦点を当てています。テクニカルノート "Single-cell T cell receptor repertoire analysis of sorted γδ T cells using the BD Rhapsody™ TCR/BCR Multiomic Assay". をご覧ください。
Immune Cell Biology
- 循環する自然リンパ球(ILC) - この研究では、シングルセル マルチオミクス解析によって特定された ILCの不均一性と、異なるドナー間における ILC サブセットを研究するためのハイパラメータ フローサイトメトリーパネルの開発に焦点を当て、独自の免疫表現型シグネチャーを明らかにしています。データシート “Resolving human circulating innate lymphoid cell heterogeneity using advanced single cell multiomic analysis.” をご覧ください。
- 制御性T細胞 (Treg) - この研究では、タンパク質とmRNAにおけるシングルセル解析について詳しく説明し、異なるTregの分化状態を明らかにしています。これは、Tregサブセットの正確な解像度を示し、免疫恒常性と疾患におけるその役割についての理解を深めています。データシート “A comprehensive characterization of regulatory T cells using BD Rhapsody™ Single-Cell Analysis System.” をご覧ください。
- 好中球 - この研究は、好中球と緊急顆粒球形成が敗血症における免疫抑制の中心であることを強調しています。シングルセル マルチオミクスにより、敗血症患者における免疫抑制性好中球の異なる集団と、変化した顆粒球形成が明らかになり、重症感染症の治療および個別化医療アプローチの潜在的なターゲットが示唆されました。.9
- CD8+T細胞 - この研究では、TetTCR-SeqHDと呼ばれる新しい手法で、TCRシーケンシングと抗原特異性、遺伝子発現、表現型解析を統合し、抗原特異的 CD8+ T細胞のプロファイルを行う方法について説明しています。このハイスループットアプローチは、抗原特異性と交差反応性を正確に検出し、1型糖尿病などの疾患におけるT細胞応答の研究に役立ちます。10
Immunotherapy
- ヒトの頭頸部扁平上皮癌(HNSCC) - HNSCC および炎症組織における免疫変化を調査したこの研究は、腫瘍特有の免疫変化が一般的な炎症パターンと並行して存在することを発見しました。 特に、腫瘍内IL1R1+制御性T (T reg) 細胞は、IL1R1- T reg細胞と比較して優れた抑制機能を示しました。T reg細胞の役割を含む腫瘍特有の免疫変化は、免疫療法戦略に影響を及ぼす可能性があります。 11
- 肺腺癌 - この研究では、EGFR変異肺腺癌における腫瘍免疫微小環境 (TME)、特にPD-L1発現の低下と腫瘍変異負荷 (TMB) の影響について説明しています。この研究では、シングルセル トランスクリプトーム解析を使用して、CD8+組織常在記憶細胞が欠如し、T細胞と他の細胞型との間のクロストークが不十分な抑制性TMEを明らかにしました。12
- 養子T細胞療法(ACT) - この研究では、がん治療におけるACTの強化について説明しています。ACTと一時的な抗CD4治療を組み合わせると、IL-18Rαhi CD8+ T細胞の存在が増加し、腫瘍抑制が改善されることを発見しました。このアプローチは免疫療法の研究を前進させる可能性があります。13
- 膵管腺癌 (PDAC) - この研究では、腫瘍微小環境内の好中球に焦点を当て、PDACにおける自然免疫の役割について説明しています。この論文では、予後不良に関連する腫瘍促進型を含む、さまざまな好中球サブグループを明らかにした研究に焦点を当てています。この研究結果は、これらの好中球を標的とすることでPDACに対する免疫療法を強化し、T細胞ベースの治療の代替手段を提供できる可能性があることを示唆しています。14
Neuroscience
- 胎児の脳の発達 - この研究は、胎児の脳の発達におけるミクログリア細胞の役割に焦点を当てています。この研究により、胚性ミクログリアが重要な皮質境界に蓄積し、形態形成ストレスによって引き起こされる空洞性病変を防ぐことが明らかになりました。この研究は、ミクログリア細胞の保護機能と、脳の形態形成中に構造的完全性を維持する上での ATM因子Spp1の重要性を強調しています。 15
- 神経ペプチド Y (NPY) - この論文では、免疫反応の調節におけるNPYの役割について説明しています。この論文では、副腎神経節と腹腔神経節のニューロンによって生成されるNPYが、脾臓の免疫活動をどのように調節できるかを強調しています。この研究により、NPYがリンパ球の増殖と活性化に影響を与えることが明らかになり、炎症性疾患や自己免疫疾患の治療に効果がある可能性が示唆されました。このニューロンと免疫システムの相互作用は、免疫バランスを維持するための古くからのメカニズムです。16
- FGF21とグルタミン酸作動性ニューロンの相互作用 - この研究では、肝臓で生成されるホルモン FGF21と、視床下部のグルタミン酸作動性ニューロンに作用して糖の摂取と嗜好を抑制するその役割について説明しています。この研究では、FGF21が視床下部腹内側部 (VMH) に直接作用して神経活動に影響を及ぼし、特に非栄養性の甘味嗜好、体重、エネルギー消費に影響を与えることなく、ショ糖摂取量を調節することが明らかになりました。17
- アルツハイマー病 (AD) - この論文では、マウス モデルにおけるABI3遺伝子のAD病理への影響について説明しています。ABI3の欠失によりアミロイドβ (Aβ) の蓄積が増加し、ミクログリア細胞の機能が損なわれ、神経炎症の悪化とシナプス機能障害を引き起こすことが明らかになりました。これは、ABI3がADの進行に役割を果たし、治療標的としての可能性を示唆しています。18
Plant Sciences
- Floral transition - The study identifies three stages of floral transition, highlights the erratic flowering in woody fruit crops like litchi due to environmental factors, and uses snRNA-seq to reveal gene expression patterns crucial for understanding bud development and flowering 19
- Organ regeneration - The study reveals that WOX5/7 proteins enhance auxin production and cytokinin sensitivity, crucial for callus pluripotency and organ regeneration. Single-cell RNA sequencing identifies key cell layers and gene interactions in Arabidopsis callus, highlighting the middle cell layer's role in shoot and root regeneration through WOX5/7 and PLT1/2 interactions.20
- Inflorescence-to-floret transition- This study profiles 37,571 rice inflorescence cells using single-cell RNA sequencing, identifying key regulators like DWARF, TILLER1 and OsAUX1. It provides insights into axillary meristem differentiation and floret development, crucial for understanding rice yield determinants. 21
- Nuclei isolation from plant samples : This study demonstrates high-efficiency nucleus isolation and purification methods for ten plant species, verified by flow cytometry. It introduces a precise absolute nucleus counting method using Trucount beads and flow cytometry, and confirms the high quality of nuclear RNA and DNA for advanced molecular research, including single nucleus RNA-seq. 22

- Li S, Simoni Y, Becht E, Loh CY, Li N et al., Human tumor-infiltrating MAIT cells display hallmarks of bacterial antigen recognition in colorectal cancer. Cell Rep Med 2020; 23(1)3:100039 doi: 10.1016/j.xcrm.2020.100039
- Ferreira R, Dopico XC, Oliveira JJ, Rainbow DB, Yang JH et al., Chronic immune activation in systemic lupus erythematosus and the autoimmune PTPN22 Trp620 risk allele drive the expansion of FOXP3+ regulatory T cells and PD-1 expression” found that SLE patients have more CD4+FOXP3+ cells due to thymically-derived Tregs expansion linked to PD-1, IL-2, and soluble SIGLEC-11. Frontiers in Immunol 2019; 10:2606 https://doi.org/10.3389/fimmu.2019.02606
- Alivernini S, MacDonald L, Elmesmari A, Finlay S, Tolusso B, et al. Distinct synovial tissue macrophage subsets regulate inflammation and remission in rheumatoid arthritis. Nat Med 2020; 8:1295-1306. doi: 10.1038/s41591-020-0939-8. Epub 2020 Jun 29
- Wang Y, Guo L, Yin X, McCarthy EC, et al. Pathogenic TNF-α drives peripheral nerve inflammation in an Aire-deficient model of autoimmunity. Proc Natl Acad Sci USA 2022; 119(4) e2114406119. doi: 10.1073/pnas.2114406119.
- Okamura T, Hamaguchi M, Tominaga H, Kitagawa N, Hoshimoto Y, et al. Characterization of peripheral blood TCR in patients with Type 1 Diabetes Mellitus by BD Rhapsody™ VDJ CDR3 assay. Cells 2022; 11(10):1623. https://doi.org/10.3390/cells11101623
- Rao Y, Zhong D, Qiu K, Cheng D, Li L, et al. Single-cell transcriptome profiling identifies phagocytosis-related dual-feature cells in a model of acute otitis media in rats. Front. Immunol. 2021; 12. https://doi.org/10.3389/fimmu.2021.760954
- May L, Chu C and Zielinkski CE. Single-Cell RNA Sequencing Reveals HIF1A as a Severity-Sensitive Immunological Scar in Circulating Monocytes of Convalescent Comorbidity-Free COVID-19 Patients. Cells; 2024; 12(4):300. https://doi.org/10.3390/cells13040300
- Li R, Li X, Zhao J, Meng F, Yao C, et al. Mitochondrial STAT3 exacerbates LPS-induced sepsis by driving CPT1a-mediated fatty acid oxidation. Theranostics; 2022; 12(2):976-998. doi: 10.7150/thno.63751
- Kwok AJ, Allcock A, Ferreira RC, Canco-Gamez E, Smee M, et al. Neutrophils and emergency granulopoiesis drive immune suppression and an extreme response endotype during sepsis. Nat Immunol. 2023; 24: 767-779 https://doi.org/10.1038/s41590-023-01490-5
- Ma K, Schonnesen A, He C, Cis AY, Sun E, et al. High-throughput and high-dimensional single-cell analysis of antigen-specific CD8+ T cells. Nat Immunol.; 2021; 22; 1590-1598 https://doi.org/10.1038/s41590-021-01073-2
- Mair F, Erickson JR, Frutoso M, Konecny AJ, Greene E, et al. Extricating human tumour immune alterations from tissue inflammation. Nature; 2022; 605(7911):728-735. https://doi.org/10.1038/s41586-022-04718-w
- Yang L, He Y-T, Dong S, Wei XX-U, Chen Z-H, et al. Single-cell transcriptome analysis revealed a suppressive tumor immune microenvironment in EGFR mutant lung adenocarcinoma. J Immunother Cancer; 2022; 10(2):e003534.
- Kim S, Cho E, Kim Y, Han C, Choi BK, et al. Adoptive immunotherapy with transient anti-CD4 treatment enhances anti-tumor response by increasing IL-18Rαhi CD8+ T cells. Nature Comm; 2022; 5314 https://doi.org/10.1038/s41467-021-25559-7
- Wang L, Liu Y, Dai Y, Tang X, Tong Y, et al. Single-cell RNA-seq analysis reveals BHLHE40-driven pro-tumour neutrophils with hyperactivated glycolysis in pancreatic tumour microenvironment. Gut; 2023; 72(5):958-971.
- Lawrence A, Canzi A, Bridlance C, Olivie N, Lansonneur C, et al. Microglia maintain structural integrity during fetal brain morphogenesis. Cell; 2024; 187(4):962-980.e19. https://doi.org/10.1016/j.cell.2024.01.012
- Yu J, Xiao K, Chen X, Hu J, Fu Z, et al. Neuron-derived neuropeptide Y fine-tunes the splenic immune responses. Neuron; 2022; 110(8):P1327-1339. https://doi.org/10.1016/j.neuron.2022.01.010
- Jensen_cody S, Flippo KH, Claflin KE, Yavuz Y, Sapouckey SA, et al. FGF21 Signals to Glutamatergic Neurons in the Ventromedial Hypothalamus to Suppress Carbohydrate Intake. Cell Metab.; 2020; 32(2):273-286. E6. Doi: 10.1016/j.cmet.2020.06.008
- Karahan H, Smith DC, Kim B, Dabin LC, Manun Al-Amin Md, et al. Deletion of Abi3 gene locus exacerbates neuropathological features of Alzheimer’s disease in a mouse model of Aβ amyloidosis. Sci Adv. ; 2021; 7(45):eabe3954. doi: 10.1126/sciadv.abe3954
- Yang, M. C., Wu, Z. C., Chen, R. Y., Abbas, F., Hu, G. B., Huang, X. M., Guan, W. S., Xu, Y. S., & Wang, H. C. (2023). Single-nucleus RNA sequencing and mRNA hybridization indicate key bud events and LcFT1 and LcTFL1-2 mRNA transportability during floral transition in litchi. Journal of Experimental Botany, 74(12), 3613-3629. https://doi.org/10.1093/jxb/erad103
- Zhai, N., & Xu, L. (2021). Pluripotency acquisition in the middle cell layer of callus is required for organ regeneration. Nature Plants, 7(1453–1460). https://doi.org/10.1038/s41477-021-01015-8
- Zong, J., Wang, L., Zhu, L., Bian, L., Zhang, B., Chen, X., Huang, G., Zhang, X., Fan, J., Cao, L., Coupland, G., Liang, W., Zhang, D., & Yuan, Z. (2022). A rice single cell transcriptomic atlas defines the developmental trajectories of rice floret and inflorescence meristems. *New Phytologist*. https://doi.org/10.1111/nph.18008
- Yang, M.-C., Wu, Z.-C., Huang, L.-L., Abbas, F., & Wang, H.-C. (2022). Systematic methods for isolating high purity nuclei from ten important plants for omics interrogation. Cells, 11(23), 3919. https://doi.org/10.3390/cells11233919
熟練した研究者であっても、この分野の新人であっても、シングルセル マルチオミクス実験に着手するには、慎重な計画、細心の注意を払った実行、そしてマルチオミクス解析の複雑さに対する深い理解が必要です。
研究テーマと実験デザイン
最初のステップは、研究課題または仮説を明確に定義することです。
シングルセル マルチオミクス解析を用いて細胞生物学や疾患病理学のどのような側面を研究することに興味がありますか? 新しい細胞タイプを特定したり、細胞状態を特徴付けたり、分子メカニズムを解明したりすることを目指していますか? 望む結果を得るためには、いくつのサンプル/セルをプロファイリングする必要がありますか? 各細胞をどの程度のシーケンス深度までシーケンスする必要がありますか? たとえば、サンプル内のサブポピュレーションを特徴付けるには、必要な細胞の数は少なくなりますが、希少な細胞を検出したり、系統を追跡したりするには、より多くの細胞が必要になります。研究目的を事前に明確にすることで、実験設計とデータ解析戦略を立て、科学的目標に効率的に取り組めるようになります。
Satija研究室のWebサイトでは、各タイプの細胞を少なくとも「n」個確認できるように、シーケンスする必要がある細胞の数を見積もるツールが提供されています。.1
実験プラットフォームを選択する
シングルセル マルチオミクス実験には通常、ゲノミクス、トランスクリプトミクス、エピゲノミクス、プロテオミクス、メタボロミクスなどの複数のオミクス手法の統合が含まれます。さまざまなテクノロジーから選択することもできますが、それぞれに長所と短所があります。研究課題と利用可能なリソースに応じて、各オミクス層に適切な実験プラットフォームとテクノロジーを選択する必要があります。実験プラットフォームを選択する際には、感度、スループット、コスト、作業時間、下流の分析ツールとの互換性などの要素を考慮してください。
BD Rhapsody™ HT シングルセル解析システムは、視覚的なワークフローQCシステムと組み合わせたマイクロウェルベースのシングルセル分割システムを提供します。

サンプルの準備と保存
実験設計に応じて、組織、細胞培養、または臨床研究サンプルからシングルセルを分離する必要がある場合があります。手動または自動の細胞分離技術を選択できます。蛍光活性化細胞選別 (FACS)、マイクロ流体細胞選別、または単純な洗浄とフィルターを使用して、サンプルを濃縮またはクリーンアップする必要がある場合があります。凍結保存、4℃保存、固定などの技術を使用してサンプルを保存する必要もあるかもしれません。結果の再現性と正確性を確保するには、サンプル準備プロトコールと品質管理対策に細心の注意を払うことが不可欠です。
データ分析と統合
シングルセル マルチオミクスデータ解析では、関係するデータセットの複雑さと次元性により、特有の計算上の課題が生じます。専門知識とリソースに応じて、バイオインフォマティクス解析を研究室内で実行するか、市販の計算プラットフォームと連携するかを選択できます。データ解析の主なステップには、品質管理、正規化、次元削減、クラスタリング、および生物学的洞察とパターンを特定するためのオミクス データセットの統合が含まれます。
BD Rhapsody™ Sequence Analysis Piplineツールの、データ解析ページにて役立つリソースをご案内しています。
解釈と検証
シングルセル マルチオミクスデータを解析したら、研究課題と既存の文献の文脈で結果を解釈します。解析からどのような生物学的知見が得られましたか? 結果は既存の仮説を支持するものでしょうか、それとも反論するものでしょうか? 結論を出す前に、研究結果を批判的に評価し、別の説明を検討することが重要です。さらに、空間マルチオミクス、qPCR、FACS、機能アッセイなどの直交技術を使用した実験検証は、重要な発見を検証し、結果の堅牢性を強化するのに役立ちます。
弊社のデータ解析ブログで、シングルセル マルチオミクスデータを処理、正規化、解釈、検証する方法について詳しく掲載しています。
- How many cells do we need to sample so that we see at least n cells of each type? Satija Lab website; accessed July 19, 2024.
研究用です。診断または治療には使用できません。